tantin
Потребител-
Брой отговори
6567 -
Регистрация
-
Последен вход
-
Days Won
27
Content Type
Профили
Форуми
Библиотека
Articles
Блогове
ВСИЧКО ПУБЛИКУВАНО ОТ tantin
-
Голямата база е с това име: v54.1.p1_1240K_public aadr_v54.1.p1_1240K_public 1240K са броя на сниповете, 1240 000 . Съответно може да ползваш и някоя по-стара. Тия най-последните версии са доста раздути. Имат много дупликати и тройно-повтарящи се. Каква полза да има 15 000 древни индивида, като много от тях ги има в 3 версии. Тоест реално са само 5000 и другите са повторения. Но да речем че повтарящите и три-пъти повтарящите се не са чак толкоз много.. Реално данните са същите и по стартите бази, просто в новите версии са включени много нови изследвания. Това обаче прави базата огрмна и трудна за обработка.
-
Забележи следното: ти тука ползваш базата: _HO_public Тази база не е с 1240к, а е само с 500к . Ако ползваш голямата база 1240к то Turkey_Kumtepe_N е само на 10%. Друг е въпроса дали тези 126 к се проектират в областта на 500к или извън тях. За предпочитане да ползваш голямата база, така има по-голям шанс да имаш покритие по същите снипове. HO_public се ползва основно за съвременни индивиди, където имаме предостатъчно покритие и информация. За древните индивиди за предпочитане да се ползва голямата база.
-
В другия файл към същата публикация са дали анализ, горе долу пълен анализ: https://www.cell.com/current-biology/pdfExtended/S0960-9822(15)01516-X Резултатите им потвърждават че тоя е много сходен с Щутгартския фермер и Айс-мен. (ледения човек от Алпите). Единствената разлика дето показват те при много-компонентния анализ е че тоя има по-голям кавказки компонент.
-
Този го нямям в ПСА базата ми. Не се е класирал, явно е с ниско покритие. Доколкото гледам в публикацията за него е някакъв типичен анатолиец (анатолийка, щото е жена) от 5 хилядолетие пне. https://www.cell.com/current-biology/pdfExtended/S0960-9822(16)30850-8 Евентуално с Ф4 статистиките може да се види спрямо кои анатолийци е най-близък, но така или иначе аз не очаквам нещо особено от такъв индивид.
-
Анотационните файлове са така, копи пействали са от различни източници. А като стане един огромен файл никой не го проверява повече.
-
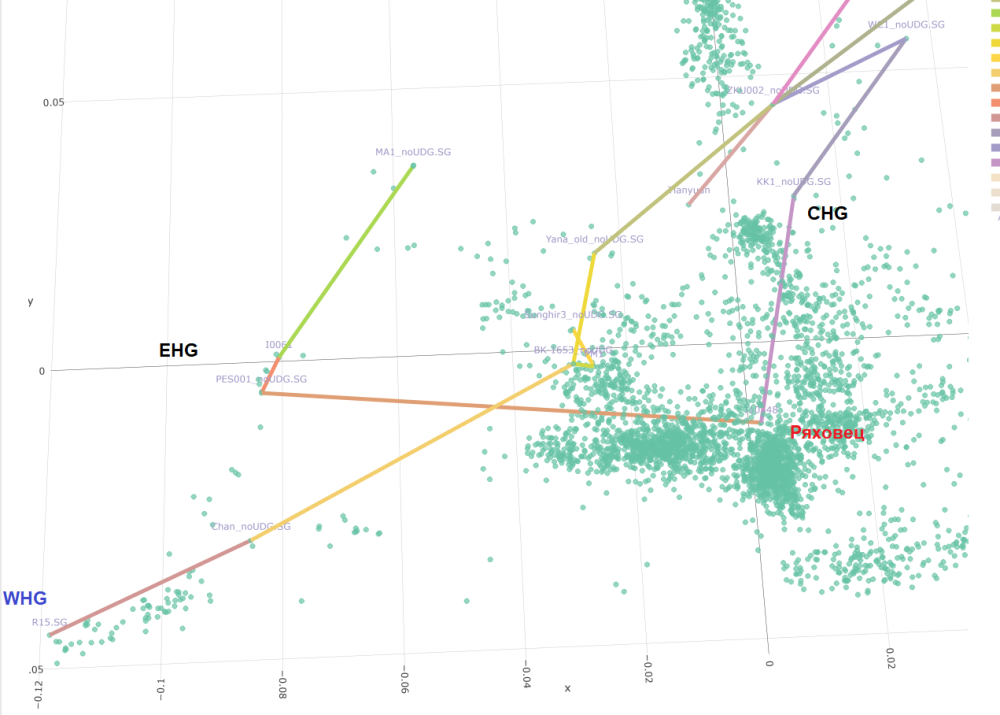
Как го тълкуваш това? EHG е малко повече от CHG ?
-
За сравнение: > extract_f2(prefix, my_f2_dir, + inds = NULL, + pops = mypops, + allsnps = TRUE, + blgsize = 0.05, + maxmem = 8000, + maxmiss = 0, + minmaf = 0, + maxmaf = 0.5, + pops2 = NULL, + outpop = NULL, + overwrite =T) i Reading allele frequencies from packedancestrymap files... i v54.1_1240K_public.geno has 16466 samples and 1233013 SNPs i Calculating allele frequencies from 177 samples in 16 populations i Expected size of allele frequency data: 296 MB 1233k SNPs read... √ 1233013 SNPs read in total ! 31339 SNPs remain after filtering. 28723 are polymorphic. i Allele frequency matrix for 31339 SNPs and 16 populations is 6 MB i Computing pairwise f2 for all SNPs and population pairs requires 201 MB RAM without splitting i Computing without splitting since 201 < 8000 (maxmem)... i Data written to D:\temp/ > mypops [1] "CHG" "EHG" "IRN_Ganj_Dareh_N" "ISR_Natufian_EpiP" "Levant_PPN" [6] "MAR_Taforalt_EpiP" "Mbuti.DG" "Mesopotamia" "RUS_AfontovaGora3" "RUS_MA1_HG" [11] "SRB_Iron_Gates_HG" "TUR_C_Boncuklu_PPN" "TUR_Marmara_Barcin_N" "TUR_Pinarbasi_EpiP" "WHG" [16] "I10548" pops_SA_right [1] "IRN_Ganj_Dareh_N" "ISR_Natufian_EpiP" "MAR_Taforalt_EpiP" "Mbuti.DG" "Mesopotamia" "RUS_AfontovaGora3" [7] "RUS_MA1_HG" "TUR_C_Boncuklu_PPN" "TUR_Pinarbasi_EpiP" "WHG" > pops_SA_left [1] "CHG" "EHG" "Levant_PPN" "TUR_Marmara_Barcin_N" "SRB_Iron_Gates_HG" > results = qpadm (f2_blocks , pops_SA_left, pops_SA_right, "I10548" ) i Computing f4 stats... i Computing admixture weights... i Computing standard errors... i Computing number of admixture waves... > results$weights # A tibble: 5 x 5 target left weight se z <chr> <chr> <dbl> <dbl> <dbl> 1 I10548 CHG 0.164 0.114 1.44 2 I10548 EHG 0.231 0.0705 3.28 3 I10548 Levant_PPN 0.0895 0.433 0.207 4 I10548 TUR_Marmara_Barcin_N 0.483 0.350 1.38 5 I10548 SRB_Iron_Gates_HG 0.0319 0.0751 0.424
-
Давам ти го Ряховеца на ПСА: Виждаш го къде е набутан в средата, в калабалъка.. И двата компонента КЛС /ИеЛС са доста силни. Най-вероятно ИеЛС е по-силния. (понеже това представяне е силно разтегнато по отношение на ЗЕЛС.
-
По принцип Лазаридис си знае работата, но и грешки стават. Възможно да е окастрил много сниповете (според извадката) . Или пък в бързината нещо да са претупали. Мога и аз да превъртя същия тест с моите данни. Това какво ни дава или променя?
-
Като гледам колко бързо напредваш, скоро може да се включваш в международния форум или да питаш директно Лазаридис. Аз също бих го тълкувал както казваш ти. Какъв е тоя от Ряховец? Ямненец вероятно?
-
При тези модели това се случва много често. Разглеждаш го като вектори. Ряховеца е много близък до ТМБ и Левант. КХГ ЕХГ се ползват за корекция с обратен знак, един вид свалят излишни компоненти.
-
Има дори излишък на информация. Стига да ти се занимава виждаш как е . Бъркаш в кацата с мед и вадиш каквото и колкото си искаш... Относно Давидски: мисля че ползва СмартПСА, обаче аз никога досега не съм успял да си го пусна тоя софтуер, обаче си ползвам друг начин за генериране на пса. Вече ПСА ни дава много удобства, видимост и всичко останало. Всичките екстри дето Давидски ги предлага идват от неговата ПСА база. А той понеже успява да "закрепи" основните компоненти, това му позволява да наблъсква в базата си колкото ще индивида. Може и 50 000 вече да ги е докарал на бройка. Но по-вероятно да е на около 20 000. Това закрепване обаче на принципните компоненти си идва с други минуси, получават се натрупвания на грешки и неточност. Засега не се напъвай още да тълкуваш сложните казуси. Понаправи си някое друго упражнение с примерите дето ги дават по уъркшоповете, вкарвай в тях отвреме на време по някоя нова тестова популация и ще се ориентираш, ще добиеш сетивата в тая сложна и хлъзгава материя.. Между другото R-studioto гърми ако му зададеш много популации, или трябва да си на много мощен компютър с много памет. Ако нещо видиш разни странни грешки: рестартираш R-studioto, намаляш малко броя на популациите и на следващото изпълнение всичко може да си е наред. За тия цели с Адмикстоолс2 R studio на Уиндоус си е достатъчно. Но за разни други неща си трябва и на Линукс.
-
-
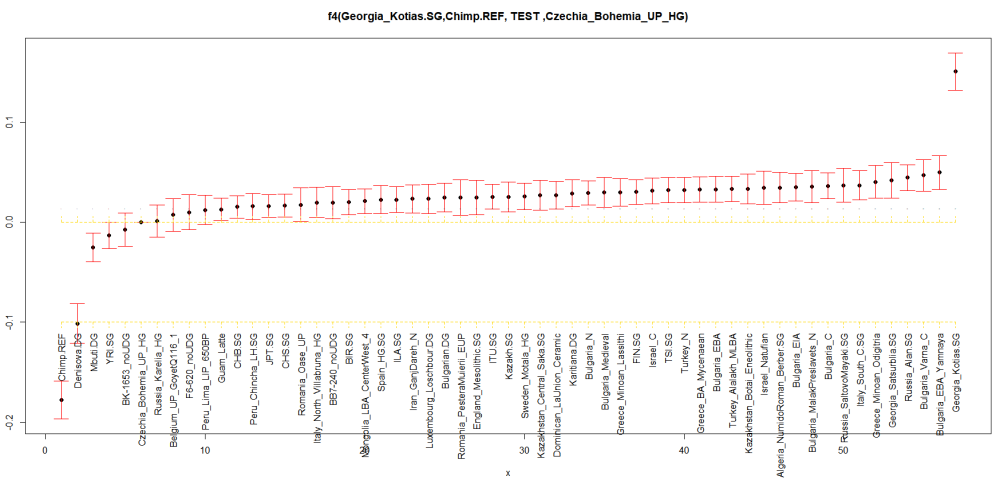
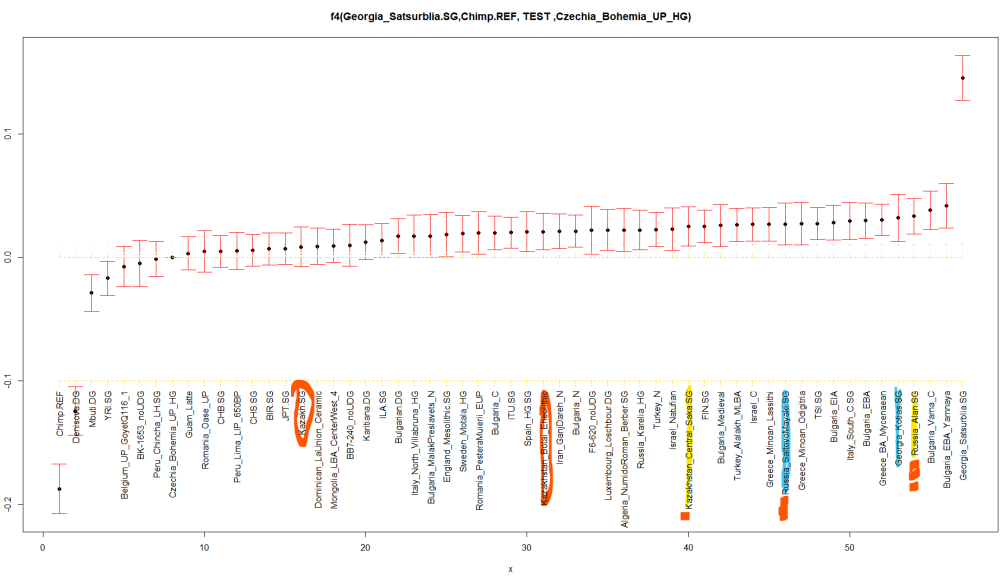
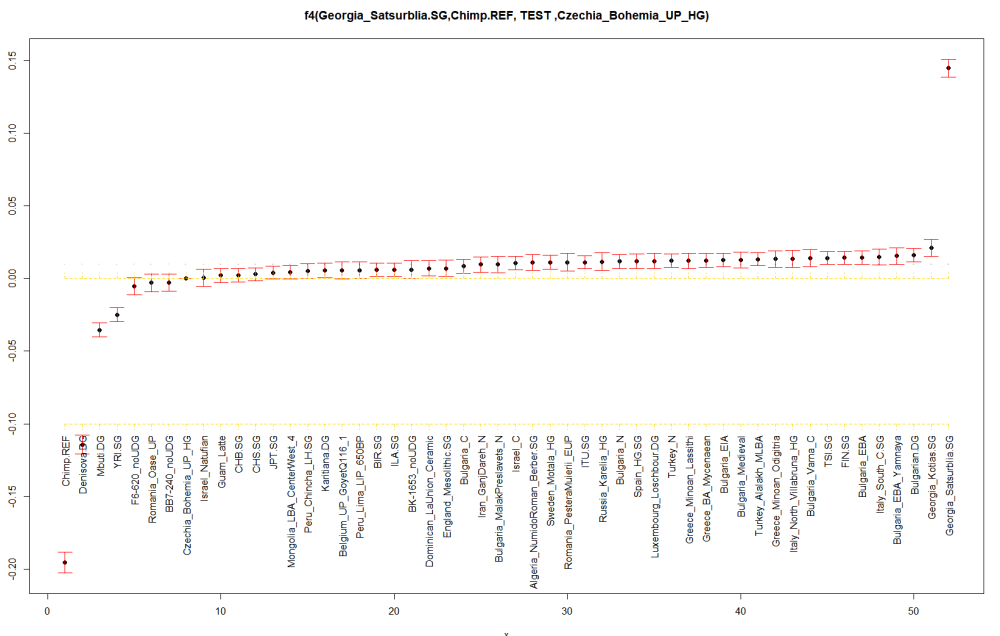
Слагам за последно и тази графика: Вместо Сацубрила, ползваме другия кавказки ловец с кодовото име КК1. (почти като куклус клан, само дето е с някакво грузинско име дето трудно може да бъде изказано). Но по-интересното е че нашите български проби показват от най-високите нива на CHG.. Включително Варна.
-
Заради теста и заради големия интерес към аланите, направих ви още един репорт, справка: Разяснителни бележки:
-
-
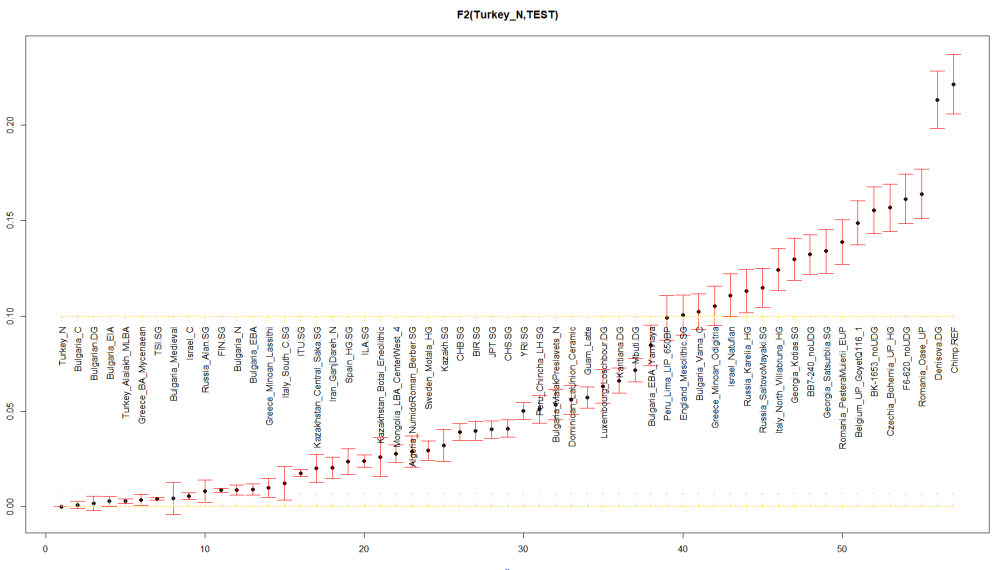
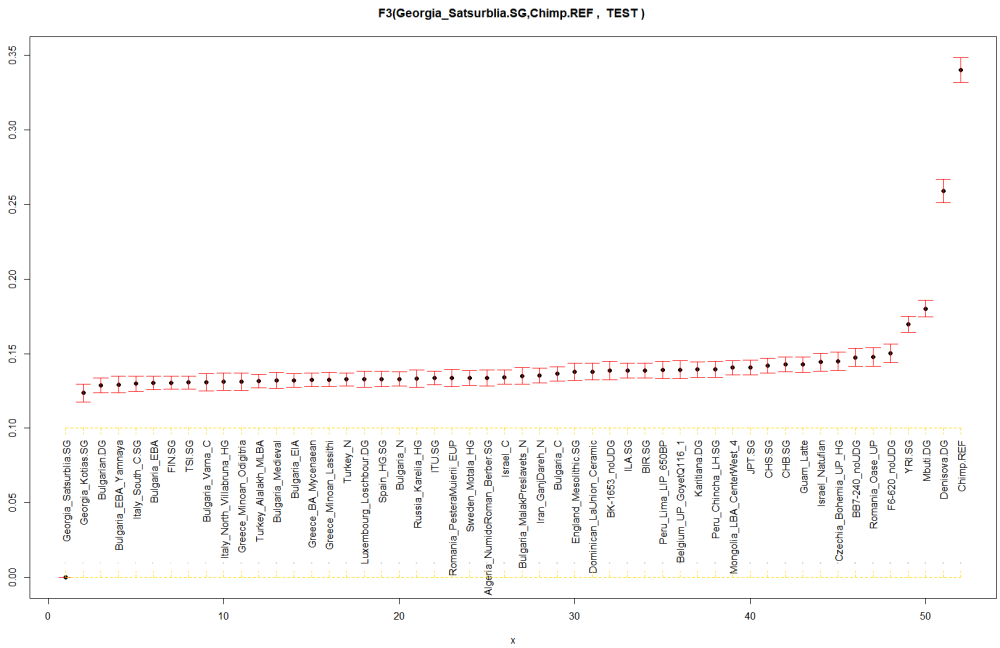
Ето и аз да дам един пример с CHG. След филтрирането ни остават едвам 5800 снипа, но и това си е ОК за целта. Все пак това са 1066 индивида в 51 групи . Резултати:
-
Дотук много добре. Щом си подкарал и Ф3 вече имаш почти всичко. Относно тълкуването: Ф3(а,в,с) равно на (а-с)(в-с) а - шимпанзето в- Кавказкия ловец с - тия дето тестваме. По принцип колкото по в средата е "с" токова по-отрицателно трябва да стане ф3. Обаче при теб вече са над нулата, тоест вече са се отклонили от средата. Тоя с по- близкото до 0 е съответно по-близък до CHG. Тоест руските алани имат малко повечко Клс компонент.
-
.. сега забелязах нещо.. това вкарване вътре на c(pop3, pop4) - функция във функция може да не работи добре. Особено ако си пробвал по същия начин с Ф3.. За предпочитане да си ги сложиш в друга променлива и да ползваш новата променлива наместо функцията. Примерно: pop_34 = c(pop3, pop4) fst(prefix, pop1, pop_34, adjust_pseudohaploid = FALSE) Най-вероятно затова Ф3 не е тръгнало.
-
Ето примерно как се прави изчисляването на тия precomp данни: # първо задаваш си къде е директорията: my_f2_dir = "C:/Users/Кухулин_123" # после правиш самото изчисление на precomp данни с екстракт функцията: extract_f2(prefix, my_f2_dir, inds = NULL, pops = mypops, blgsize = 0.05, maxmem = 8000, maxmiss = 0, minmaf = 0, maxmaf = 0.5, pops2 = NULL, outpop = NULL, overwrite =T) # Тука mypops = pop1 + pop2 + pop3 + pop4 - всичките трябва да ги има в тоя списък # после ги четеш от директорията: f2_blocks = f2_from_precomp(my_f2_dir, pops = mypops) # И после директно с тях си изчисляваш: f4(f2_blocks, pop1, pop2, pop3, pop4 )
-
За утре имаш следната задача: Вместо това: prefix f4(prefix, pop1, "Chimp.REF", pop3, pop4) Ще ползваш f2_from_precomp Обаче трябва малко подготовка, да си подготвиш един фолдер дето да се запишат прочетените данни и ти оттам да ги ползваш наготово , а не всеки път да ги смяташ на ново и наново.. Има го добре обяснено в оня workshop. f2_blocks = f2_from_precomp(my_f2_dir, pops = mypops) вече като подкараш Ф4 с опцията f4(f2_blocks, pop1, pop2, pop3 ) ще видиш как стават другите неща.
-
Едва ли.. При толкоз включени снипове: Computing block lengths for 1150639 SNPs... Но да.. Вероятно си прав. Не знам точно какво търсиш или какво очакваш . Близост или раздалечаване? Не забравяй че имаш и Ф3, която дори е по-лесна от Ф4. С Ф3 по-лесно можеш да докажеш близост, или кой е по средата. Зет се изчислява по същия начин. f3(prefix, pop1, pop2 , pop3)
-
За момента не го мисли това със z, всяко нещо с времето си. Z мисля че го изчисляваха като z= est/se . За днес това ти е предостатъчно. Запази някоя друга мозъчна клетка за утре.
-
Сега бачо Кухулине, слушай внимателно: взимаш си файла: v54.1.p1_1240K_public.ind / да речем че с тая база работиш. Отваряш го с ноутпад.. И си избираш кой индивид искаш да провериш. да речем за тия индивиди: DA160_noUDG.SG F Russia_Alan.SG DA162_noUDG.SG M Russia_Alan.SG DA164_noUDG.SG F Russia_Alan.SG DA243_noUDG.SG M Russia_Alan.SG Намираш ги във файла ти къде са: и ги заместваш по тоя начин: DA160_noUDG.SG F DA160_noUDG.SG DA162_noUDG.SG M DA162_noUDG.SG DA164_noUDG.SG F DA164_noUDG.SG DA243_noUDG.SG M DA243_noUDG.SG веднъж като го презапишеш тоя файл, (save), мошеж да си правиш проверката директно по индивид. Няма нужда да ползваш груповото име Russia_Alan.SG , а ползваш индивидуланото име за съответния индивид. Правиш си бакъп на оригиналния v54.1.p1_1240K_public.ind ако решиш да го ползваш пак с групата вместо по индивидите.
-
F4 статистиките работят само с действителни снипове, където тия снипове са представени при всичките тествани популации. Ако липсват сниповете - то се прави филтриране на липсващите снипове и се махат същите от всичките тествани популации. Докато Г25 и разни други методи правят изчисленията на базата дистанции. И после ти показват апроксимации. Примерно само на базата на 1-ва хромозома ти можеш да дадеш приблизителна оценка къде е позицията на индивида по всичките 22 хромозоми. И при това едва ли ще имаш грешка в повече от 20 %.. Ф4 статистиките ни позволяват да си правим тестове с много окастрени данни, да сравним тия окастрени данни с окастрените данни от другите групи и да преценим взаимното им разположение едни спрямо други.